Gli schemi di macchie ottenuti da soluzioni peptidiche essiccate consentono un'identificazione efficace delle malattie neurodegenerative, un'analisi rapida con reti neurali.
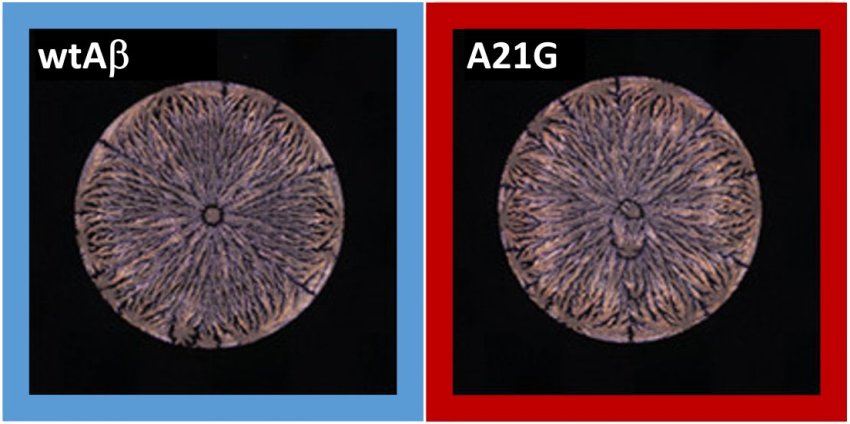 Le reti neurali possono rilevare differenze minime nei modelli di colorazione dalle soluzioni di peptidi essiccate (a sinistra: peptide Aβ42 - a destra: mutazione).
Le reti neurali possono rilevare differenze minime nei modelli di colorazione dalle soluzioni di peptidi essiccate (a sinistra: peptide Aβ42 - a destra: mutazione).
Le malattie neurodegenerative come il morbo di Alzheimer (MA) o il Parkinson sono causate da errori di piegatura (misfolding o ripiegamento errato) di proteine o peptidi, cioè da cambiamenti nella loro struttura spaziale. Questo è il risultato di minuscole deviazioni nella composizione chimica delle biomolecole.
Ricercatori del Karlsruhe Institute of Technology (KIT) hanno sviluppato un metodo semplice ed efficace per rilevare tale errata piegatura in una fase iniziale della malattia. Il misfolding è rivelato dalla struttura dei residui secchi di soluzioni proteiche e peptidiche. Il metodo prevede l'analisi delle micrografie con reti neurali e ha una precisione predittiva di oltre il 99%. I risultati sono stati pubblicati su Advanced Materials.
La struttura biochimica di proteine e peptidi determina le loro funzioni biologiche. Ci sono molte indicazioni che anche minuscoli cambiamenti strutturali o spaziali possano promuovere lo sviluppo delle malattie. Molte malattie neurodegenerative sono state attribuite al ripiegamento errato di proteine e peptidi causati da tali cambiamenti. I peptidi di amiloide-beta (Aβ42) hanno un ruolo chiave nel MA; differiscono in un singolo residuo di aminoacidi e rappresentano mutanti ereditari della malattia.
Finora non c'era un metodo semplice e accurato per prevedere le mutazioni nelle proteine. Al KIT, un gruppo di ricerca guidato dal professor Jörg Lahann ha sviluppato un metodo per rilevare il ripiegamento errato attraverso la struttura delle soluzioni di proteine e peptidi essiccati.
"I modelli di macchiatura non erano solo caratteristici e riproducibili, ma si traducono anche in una classificazione di 8 mutazioni con una precisione predittiva di oltre il 99%", ha affermato Lahann, autore senior dello studio, nel descrivere i risultati.
Il gruppo ha dimostrato che le informazioni cruciali sulle strutture primarie e secondarie dei peptidi possono essere raccolte dalle macchie lasciate indietro mediante goccioline in essiccazione della soluzione peptidica su una superficie solida.
Modelli di macchiatura come impronte digitali del peptide
Le soluzioni proteiche e peptidiche sono posizionate con precisione su vetrini da un sistema di pipettatura automatizzato per garantire risultati controllati e riproducibili. Le superfici dei vetrini sono preparate in anticipo con un rivestimento polimerico idrofobico.
Per analizzare i modelli di colorazione complessi delle goccioline essiccate, i ricercatori hanno acquisito immagini della microscopia di polarizzazione. Le immagini sono state quindi analizzate con reti neurali ad apprendimento profondo.
"Poiché le strutture sono molto simili e difficili da distinguere a occhio nudo, è stata sicuramente una sorpresa che le reti neurali fossero così efficaci", afferma Lahann a proposito dei risultati. "I modelli di macchiatura dei peptidi Aβ servono da impronte digitali esatte che riflettono l'identità strutturale e spaziale di un peptide".
Questa tecnologia consente di identificare in pochi minuti le varianti di MA con la massima risoluzione, secondo Lahann.
La preparazione semplice del campione offre diagnosi veloci
I risultati suggeriscono che un metodo semplice come essiccare una goccia di soluzione peptidica su una superficie solida può diventare un indicatore per differenze minime nelle strutture primarie e secondarie dei peptidi.
"Sono urgentemente necessari metodi di rilevamento scalabili e accurati per stratificare le alterazioni conformazionali e strutturali delle proteine, per decodificare le firme patologiche di malattie come Il MA e il Parkinson", afferma Lahann.
È anche un metodo relativamente semplice che non richiede una preparazione elaborata di campioni e quindi consente una diagnosi semplice e amichevole per i pazienti. Inoltre, il metodo ha un grande potenziale per altre applicazioni nella diagnostica medica e nella rilevazione molecolare delle malattie.
Fonte: KIT-Karlsruher Institut für Technologie (> English) - Traduzione di Franco Pellizzari.
Riferimenti: Azam Jeihanipour, Jörg Lahann. Deep‐Learning‐Assisted Stratification of Amyloid Beta Mutants Using Drying Droplet Patterns. Advanced Materials, 2022, DOI
Copyright: Tutti i diritti di testi o marchi inclusi nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non rappresenta necessariamente l'opinione dell'Associazione Alzheimer OdV di Riese Pio X ma solo quella dell'autore citato come "Fonte". I siti terzi raggiungibili da eventuali collegamenti contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.





 Associazione Alzheimer OdV
Associazione Alzheimer OdV